| Autor |
Nachricht |
xpimp77
Anmeldungsdatum: 26.03.2008
Beiträge: 4
|
 Verfasst am: 26. März 2008 00:13 Titel: MCS und Transkriptionsstart Verfasst am: 26. März 2008 00:13 Titel: MCS und Transkriptionsstart |
 |
|
Hi Leute!
Wie insertiert man ein Insert in einen Plasmidvektor fachgerecht?
Muss mein Insert (mit ATG beginnend) welches ich in die MCS einsetzen will, in Frame mit dem Transkriptionsstart sein?
Oder kann mir das egal sein weil mein Gen erst mit dem ATG beginnt?
Gruß,
xpimp
|
|
 |
Bert

Anmeldungsdatum: 26.09.2007
Beiträge: 82
Wohnort: Niedersachsen
|
 Verfasst am: 26. März 2008 13:18 Titel: Re: MCS und Transkriptionsstart Verfasst am: 26. März 2008 13:18 Titel: Re: MCS und Transkriptionsstart |
 |
|
| xpimp77 hat Folgendes geschrieben: | Hi Leute!
Wie insertiert man ein Insert in einen Plasmidvektor fachgerecht?
Muss mein Insert (mit ATG beginnend) welches ich in die MCS einsetzen will, in Frame mit dem Transkriptionsstart sein?
Oder kann mir das egal sein weil mein Gen erst mit dem ATG beginnt?
Gruß,
xpimp |
Es kommt darauf an, was du machen willst.
1. Du willst einen DNA-Abschnitt sequenzieren.
2. Du willst ein Gen heterolog exprimieren.
3. Du willst etwas anderes untersuchen. Was?
_________________
Holzhacken ist deshalb so beliebt, weil man bei dieser Tätigkeit den Erfolg sofort sieht. [Albert Einstein] |
|
 |
xpimp77
Anmeldungsdatum: 26.03.2008
Beiträge: 4
|
 Verfasst am: 26. März 2008 16:36 Titel: Verfasst am: 26. März 2008 16:36 Titel: |
 |
|
Hi!
Antwort B - Ich möchte das Gen heterolog exprimieren.
Bei nem Sequenzierplasmid wärs mir ja egal ob in Frame oder nicht.
Wenn du dir ein Datenblatt von nem Plasmid anschaust z. B. pcDNA3, dann steht da in der Beschreibung der MCS ein Pfeil mit der Bezeichnung: "putative transcriptional start" also Transkriptionsstart.
Es geht mir darum ob mein Startcodon ATG im gleichen Frame wie dieser Transkriptionsstart sein muss, weil ja dort die Transkription losgeht.
Oder ist dies bei der Transkription egal, weil ja erst bei der Translation das ATG erkannt und als Startpunkt festgelegt wird.
Wird quasi erst bei der Translation der Frame festgelegt? Oder schon vorher bei der Transkription?
Bis denn,
xpimp
|
|
 |
Bert

Anmeldungsdatum: 26.09.2007
Beiträge: 82
Wohnort: Niedersachsen
|
 Verfasst am: 26. März 2008 19:00 Titel: Verfasst am: 26. März 2008 19:00 Titel: |
 |
|
| xpimp77 hat Folgendes geschrieben: | Hi!
Antwort B - Ich möchte das Gen heterolog exprimieren.
|
Für eine effektive heterologe Expression sollte „dein“ ORF mit dem Plasmid-ATG in frame sein, mehr noch, du solltest in „deinem“ Gen die Nukleotide vor dem ATG (inklusive ATG!) entfernen und mit dem Codon der ersten Aminosäure direkt hinter das Plasmid-ATG setzen (wie im Beispiel 1). Wenn der Wirt das fremdexprimierte Produkt auch in hohen Dosen verträgt, würde ich das so machen. Aber...
Generell ist es nicht auszuschließen, daß das Fremdgen-ATG von dem Wirtsapparat erkannt wird; habe ich auch schon erlebt und eine (wenn auch sehr schwache) Fremdgenexpression erreicht, obwohl das Fremdgen-ATG nicht mit dem Plasmid-ATG in frame war (Beispiel 3). Das fremdexprimierte Enzym war sogar aktiv, allerdings lag seine Aktivität bei nur etwa 20% der „normalen“ Aktivität. Dies ist manchmal erwünscht, wenn das fremdexprimierte Produkt toxisch wirkt. Natürlich wird nicht jedes Gen so (Beispiel 3) exprimiert, aber in manchen Fällen klappt es. 
Beispiel:
Das ist dein Insert (Nukleotidsequenz):
5’...GGATCCatggacgcctccatctccaccgacgagatcaacgagaactcctacatgtaa...3’
Das ist dein Peptid (Aminosäuresequenz):
N-term... D A S I S T D E I N E N S Y M…C-term
Das sind einige ExpressionsMöglichkeiten (Rot ist die Plasmid-DNA, blau ist deine Insert-DNA):
| Beschreibung: |
|
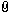
Download |
| Dateiname: |
Expression.png |
| Dateigröße: |
6.7 KB |
| Heruntergeladen: |
3866 mal |
_________________
Holzhacken ist deshalb so beliebt, weil man bei dieser Tätigkeit den Erfolg sofort sieht. [Albert Einstein] |
|
 |
xpimp77
Anmeldungsdatum: 26.03.2008
Beiträge: 4
|
 Verfasst am: 26. März 2008 19:53 Titel: Verfasst am: 26. März 2008 19:53 Titel: |
 |
|
Hi!
Erstmal tausend dank für deine Antwort.
Ich hab dir hier mal ein Bild meiner Anfangssequenz und die original pcDNA3-MCS drangehängt.
Hatte mein Insert mit HindIII und BamHI reingesetzt.
Ich finde kein Plasmid-Startcodon. Es gibt nur meins.
Erst ziemlich weit 5' ist ein ATG (dunkel markiert), ist aber Bestandteil der CAAT-Box.
Wär denk ich doof, wenn ich das alles wegschneiden würde :-).
Mein ATG ist jedoch nicht in frame mit dem AGAGAA...-Transkriptionsstart, wie es in den Unterlagen von pcDNA3 angegeben ist. Das hatte bei mir Bedenken ausgelöst.
Bis denn,
Xpimp
| Beschreibung: |
|
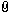
Download |
| Dateiname: |
x.jpg |
| Dateigröße: |
53.88 KB |
| Heruntergeladen: |
3869 mal |
| Beschreibung: |
|
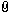
Download |
| Dateiname: |
pcDNA3.jpg |
| Dateigröße: |
69.51 KB |
| Heruntergeladen: |
3860 mal |
| Beschreibung: |
|
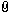
Download |
| Dateiname: |
atg.jpg |
| Dateigröße: |
52.14 KB |
| Heruntergeladen: |
3863 mal |
|
|
 |
Bert

Anmeldungsdatum: 26.09.2007
Beiträge: 82
Wohnort: Niedersachsen
|
 Verfasst am: 27. März 2008 02:00 Titel: Verfasst am: 27. März 2008 02:00 Titel: |
 |
|
| xpimp77 hat Folgendes geschrieben: |
Ich finde kein Plasmid-Startcodon. Es gibt nur meins.
Erst ziemlich weit 5' ist ein ATG (dunkel markiert), ist aber Bestandteil der CAAT-Box.
|
Das von dir markierte ATG ist nicht das Startcodon. Dein Startcodon ist hinter der HindIII-Stelle: ...5’aagcttacgATGgt...3’ und es stammt von deinem Insert. Im Plasmid ist bis zu der HindIII-Stelle keines.
Das ist also OK. Wichtig ist die Umgebung des Startcodons – das a bei –3 und das g bei +4, da es sich wohl um Expression in Säugetierzellen handelt (vermute ich mal). Das hast du auch richtig. 
Generell mußt du auf „in frame“ nur dann achten, wenn du diese Komponenten (RBS mit ATG, irgendein tag mit Stopcodon oder andere funktionelle Komponenten als Fusion usw.) aus dem Plasmid nutzt. Das scheint bei dir nicht der Fall zu sein, allerdings habe ich den 3’ nicht gesehen; falls du da eine tag-Sequenz nutzt (z.B. His-tag), so muß dein Insert mit dem C-term des Plasmids in frame sein (also mit dem His-tag und dem Stopcodon!).
_________________
Holzhacken ist deshalb so beliebt, weil man bei dieser Tätigkeit den Erfolg sofort sieht. [Albert Einstein] |
|
 |
xpimp77
Anmeldungsdatum: 26.03.2008
Beiträge: 4
|
 Verfasst am: 27. März 2008 11:15 Titel: Verfasst am: 27. März 2008 11:15 Titel: |
 |
|
Hey, Danke dir vielmals Bert!
Deine kompetenten Antworten haben mir sehr geholfen.
Vielleicht postet man sich mal wieder.
Machs gut,
Xpimp
|
|
 |
|