| Autor |
Nachricht |
jörg

Anmeldungsdatum: 12.12.2010
Beiträge: 2107
Wohnort: Bückeburg
|
 Verfasst am: 07. Nov 2011 10:15 Titel: Verfasst am: 07. Nov 2011 10:15 Titel: |
 |
|

_________________
RNA?- just another nucleic acid? |
|
 |
benno67
Gast
|
 Verfasst am: 07. Nov 2011 14:28 Titel: Verfasst am: 07. Nov 2011 14:28 Titel: |
 |
|
so also damit ich mal wieder...
die dritte abbildung versteh ich mal wieder nicht.
also wir haben ja wieder einen doppelstrang im denaturierten zustand und es soll jeweils die rote sequenz entfernt werden. hierzu werden wieder vier primer verwendet. P1 synthetisiert den grünen abschnitt bis zur roten sequenz. P3 mit seinem Überhang synthetisiert die blaue sequenz, also natürlich jeweils komplementär dazu. P2 und P4 machen das in gleicher weise. stimmt das erstmal so? aber woher weiß die polymerase denn dass sie ausgehend von den primern nur bis zur roten sequenz synthetisieren soll? und warum besitzt P3 wieder einen überhang? sind die primer wieder irgendwie komplementär? weil das kann ich nicht wirklich erkennen...
|
|
 |
jörg

Anmeldungsdatum: 12.12.2010
Beiträge: 2107
Wohnort: Bückeburg
|
 Verfasst am: 07. Nov 2011 14:59 Titel: Verfasst am: 07. Nov 2011 14:59 Titel: |
 |
|
| benno67 hat Folgendes geschrieben: | | aber woher weiß die polymerase denn dass sie ausgehend von den primern nur bis zur roten sequenz synthetisieren soll? |
Hier liegt das Geheimnis -wie eigentlich immer bei der PCR- im zweiten Zyklus:
Im ersten Zyklus wird natürlich über die rote, zu deletierende Sequenz herübergelesen. Im zweiten Zyklus allerdings bindet an den neuen, mit P1 synthetisierten Strang (der revers komplementär zur Matrize ist!!!!) ja der Primer P2, wodurch dann "das andere Ende" festgelegt wird.
Nun kann die Polymerase ja nur noch bis zu der Stelle amplifizieren, an der P1 im ersten Zyklus gebunden hat, weil da der Strang zu Ende ist.
Ich glaube, du solltest dir noch einmal die Grundlagen der PCR-Amplifikation anschauen, denn damit hätte das eigentlich klar sein müssen.
Denn wie erkennt die Polymerase überhaupt, wo die zu amplifizierende Sequenz zuende ist?
Sie beginnt an einem Primer und legt los. Dabei überschreibt sie natürlich die Stelle, an der im nächsten Zyklus auf dem antiparallelen Strang der Reverse Primer bindet.
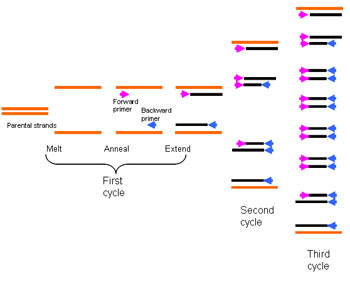
Ich habe dir mal ein Bild von der österreichischen summerschool angehängt (www.summerschool.at), das das verdeutlicht.
| benno67 hat Folgendes geschrieben: | | und warum besitzt P3 wieder einen überhang? sind die primer wieder irgendwie komplementär? |
Ja, sind sie, es wird wieder eine Fusionssequenz erzeugt, nur diesmal nicht mit Sequenzen von verschiedenen Genen, sondern von demselben.
Das Prinzip ist aber das gleiche.
| Beschreibung: |
|
| Dateigröße: |
35.49 KB |
| Angeschaut: |
9664 mal |
|
_________________
RNA?- just another nucleic acid? |
|
 |
benno67
Gast
|
 Verfasst am: 07. Nov 2011 15:46 Titel: Verfasst am: 07. Nov 2011 15:46 Titel: |
 |
|
|
also wir nehmen jetzt mal nur den unteren teilstrang. im ersten zyklus bindet P1 an den Einzelstrang und legt damit den anfangspunkt der sequenz fest. die polymerase synthetisiert dann den strang komplementär, sodass also praktisch der gleich strang nochmal entsteht (gleich farben) mit dem unterschied dass er antiparallel zum template ist. dann wird der entstandene doppelstrang wieder denaturiert und liegt als enzelstränge vor. am oberen einzelstrang lagert sich dann P2 an. aber woher weiß er dann wo die sequenz ist an die er sich anlagern muss? ist der Primer sequenzspezifisch? ausgehend von P2 synthetisiert Polymerase dann den abschnitt bis zum ende, das ja durch P1 im ersten zyklus festgelegt wurde. aber dann gibt es ja noch die andere seite neben der zu deletierenden sequenz. da müsste sich ja nun wieder P4 anlagern und polymerase ergänzt wieder. aber woher weiß sie dass dort das ende erreicht ist? eigentlich ist doch das ende erreicht wenn der nächste zyklus stattfindet oder?
|
|
 |
jörg

Anmeldungsdatum: 12.12.2010
Beiträge: 2107
Wohnort: Bückeburg
|
 Verfasst am: 07. Nov 2011 18:34 Titel: Verfasst am: 07. Nov 2011 18:34 Titel: |
 |
|
| benno67 hat Folgendes geschrieben: | | aber woher weiß er dann wo die sequenz ist an die er sich anlagern muss? ist der Primer sequenzspezifisch? |
Was ist denn eigentlich ein Primer?
| benno67 hat Folgendes geschrieben: | | aber woher weiß sie dass dort das ende erreicht ist? eigentlich ist doch das ende erreicht wenn der nächste zyklus stattfindet oder? |
Die Frage verstehe ich nicht. Das funktioniert ganz genauso wie auf der anderen Seite.
Schau dir wirklich noch einmal die Grundlagen an.
Wie sehen die Stränge einer zu amplifizierenden Sequenz nach dem ersten Zyklus aus?
Und wie nach dem zweiten?
Beschreibe bitte ganz allgemein anhand der Abbildung, die ich an den letzten Beitrag angehängt habe.
Es reicht, wenn du einen Strang durch die Zyklen begleitest, aber du solltest dich dem nach meiner Einschätzung noch einmal ganz basal nähern.
_________________
RNA?- just another nucleic acid? |
|
 |
benno67
Gast
|
 Verfasst am: 08. Nov 2011 09:35 Titel: Verfasst am: 08. Nov 2011 09:35 Titel: |
 |
|
|
ich hab dad jetzt erstmal noch eine grundlegende frage: um einen DNA-Abschitt zu vervielfältigen müssen die flankierenden Bereiche (etwa 20 bp) bekannt. zu diesen sequenzen werden dann komplementäre oligonukleotide(=Primer) hergestellt, die dann daran binden können. Der anfang und das ende der zu amplifizierenden sequenz wird durch die primer festgelegt. genau das verstehe ich nicht. die primer dienen ja als startpunkt für die polymerase. das ist klar. aber wie kann jetzt der andere primer das ende darstellen? wie kann ich mir das vorstellen? Hat das irgendwas damit zu tun dass die polymerase auch die komplementäre sequenz des primers synthetisiert? oder wie?
|
|
 |
benno67
Gast
|
 Verfasst am: 08. Nov 2011 09:48 Titel: Verfasst am: 08. Nov 2011 09:48 Titel: |
 |
|
ich hab hier noch ein bild aber das kann ich leider nicht einfügen also beschreib ich das wieder: wenn du in google pcr eingibst (bei Bilder) ist es das dritte in der ersten reihe.
und zwar sieht man ja da in der zu verviefältigten sequenz jeweils am anfang und am ende (5' und 3') noch solche kurze dunklen abschnitt die nicht zur zielsequenz gehören. was haben diese zu beudeuten und warum werden sie manchmal mitsynthetisert und manchmal nicht? das hat denk ich mal wieder was mit mit eben gestellten frage zu tun...
|
|
 |
jörg

Anmeldungsdatum: 12.12.2010
Beiträge: 2107
Wohnort: Bückeburg
|
 Verfasst am: 08. Nov 2011 10:05 Titel: Verfasst am: 08. Nov 2011 10:05 Titel: |
 |
|
Betrachten wir dazu einmal einen Strang:
An dem 3'-Ende der zu amplifizierenden Sequenz bindet ein Primer A und vervielfältigt diesen Strang von der Sequenz ausgehend, an die er bindet über die eigentliche Zielsequenz hinaus bis in "die dunklen Abschnitte" (wie du sie genannt hast).
Das Resultat ist nach dem ersten Zyklus ein komplementärer Strang, der mit der Primersequenz A am 5'-Ende beginnt. An dem anderen Ende der zu amplifizierenden Sequenz, also irgendwo "in der Mitte" des neuen Stranges, bindet nun im zweiten Zyklus Primer B ebenso am 3'-Ende des neu zu entstehenden Stranges, dieser kann jedoch nur bis zu der Stelle amplifiziert werden, an der die Primersequenz A sich befindet, da der Strang hier zuende ist.
Ausgehend von diesem einen Strang haben wir nach zwei Zyklen also drei verschieden lange Stränge:
Den Ausgangsstrang, ein Amplifikat, das an seinem 5'-Ende von Primersequenz A begrenzt wird, an dem 3'-Ende aber länger ist, als die Zielsequenz es sein soll und einen Strang, der die Zielsequenz enthält.
Ersterer bleibt zwar vorhanden, wird aber in den folgenden Zyklen nicht vollständig vervielfältigt, zweiterer vervielfältigt sich linear (je Zyklus kommt je Strang ein neuer davon hinzu, der direkt von dem Ausgangsstrang ausgehend amplifiziert wird) und die Zielsequenz vervielfältigt sich exponentiell.
_________________
RNA?- just another nucleic acid? |
|
 |
benno67
Gast
|
 Verfasst am: 08. Nov 2011 10:29 Titel: Verfasst am: 08. Nov 2011 10:29 Titel: |
 |
|
|
und was genau sind jetzt diese "dunklen Abschnitte"? aber ich versteh auch noch nicht so ganz warum die synthese des komplementären stranges bei dem anderen primer aufhört? wie muss ich mr denn das vorstellen?
|
|
 |
jörg

Anmeldungsdatum: 12.12.2010
Beiträge: 2107
Wohnort: Bückeburg
|
 Verfasst am: 08. Nov 2011 10:54 Titel: Verfasst am: 08. Nov 2011 10:54 Titel: |
 |
|
| benno67 hat Folgendes geschrieben: | | und was genau sind jetzt diese "dunklen Abschnitte"? |
Da läuft der Ausgangsstrang weiter. Sie gehören nicht zur Zielsequenz, sind aber Bestandteil der Matrize, die ja gewöhnlicherweise länger als die Zielsequenz ist: Zielsequenz z.B. 800 Basenpaare, die von einem Chromosom amplifiziert werden sollen, das über mehrere Millionen Basenpaare verfügt.
| benno67 hat Folgendes geschrieben: | | aber ich versteh auch noch nicht so ganz warum die synthese des komplementären stranges bei dem anderen primer aufhört? |
Weil der Strang da einfach nach dem ersten Zyklus zuende ist.
Noch einmal:
1. Zyklus:
Am 3'-Ende der zu vervielfältigenden Sequenz (nicht gleich 3'-Ende des Ausgangsstranges!!!) bindet ein Primer A.
An diesem beginnt die Polymerase ihre Arbeit und schreibt nun einen revers komplementären Strang, also in 5'-3'-Richtung.
Am 5'-Ende ist dieser Strang nun an der Stelle, wo der Primer gebunden hat zuende.
Ich habe nun zwei antiparallele Stränge:
Den Ausgangsstrang ("volle Länge") und einen "mittellangen" Strang, der bereits ein definiertes 5'-Ende der Zielsequenz hat.
Die Zielsequenz wir im Folgenden der Einfachheit halber "kurzer Strang" genannt.
2. Zyklus:
Mit dem "vollen-Länge-Strang" geschieht das gleiche wie im ersten Zyklus, deswegen gilt unsere Aufmerksamkeit dem "mittellangen" Strang:
Dieser ist also am 5'-Ende bereits definiert, weil da ja in Zyklus 1 der Primer A gebunden hat und die Polymerase dort ihre Arbeit begann.
Am 3'-Ende der Zielsequenz (wieder: nicht gleich dem 3'-Ende der Vorlage!!), also in der "Mitte" dieses Stranges bindet nun Primer B und von hier aus arbeitet die Polymerase auf das bereits definierte 5'-Ende zu. An diesem 5'-Ende ist der Strang dort zuende, wo im ersten zyklus der Primer A gebunden hat, also kann die Polymerase nicht weitermachen.
_________________
RNA?- just another nucleic acid? |
|
 |
benno67
Gast
|
 Verfasst am: 08. Nov 2011 13:32 Titel: Verfasst am: 08. Nov 2011 13:32 Titel: |
 |
|
ahh jetzt hab ich das verstanden 
ok dann nochmal zur 3. abbildung:
in der PCR1 werden ja nur P1 und P2 gebraucht. das heißt hierbei ist auch der erste zyklus eingeschlossen bei dem die polymerase den gesamten strang einschließtlich der roten sequenz komplementär synthetisiert. an den oberen strang (der nach rechts orientiert ist und P1 am startpunkt hatte) bindet dann P2 und synthetisiert die sequenz bis zum ende (also da wo zuvor P1 gebunden hat). dadurch erhält man doch einen doppelstrang der aus grüner sequenz (oberer teilstrang) und aus grüner sequenz mit blauen überhang (unterer teilstrang) besteht. So und erst dann wird wieder P1 benötigt oder? dieser bindet dann an den teilstrang mit dem überhang und synthetisiert den komplementären strang dazu so dass man nach PCR1 die den dargesgtellten doppelstrang erhält. ist das so richtig? also kann man doch auch sagen dass durch PCR1 der nach rechts orientierte Teilstrang bearbeitet wird und durch PCR2 der nach links orientierte teilstrang oder? achso und der überhang von P3 soll bestimmt grün sein oder?
|
|
 |
jörg

Anmeldungsdatum: 12.12.2010
Beiträge: 2107
Wohnort: Bückeburg
|
 Verfasst am: 08. Nov 2011 13:50 Titel: Verfasst am: 08. Nov 2011 13:50 Titel: |
 |
|
| benno67 hat Folgendes geschrieben: | | So und erst dann wird wieder P1 benötigt oder? |
So kannst du dir das vorstellen, ganz richtig ist es jedoch nur für den betrachteten Strang der "Vorlage". Für den komplementären Strang gilt das ganze natürlich auch, nur mit den jeweils anderen Primern, da er ja antiparallel ausgerichtet ist, also die Sequenz am 3'-Ende komplementär zu der Sequenz am 5'-Ende das gegenüberliegenden Stranges ist.
| benno67 hat Folgendes geschrieben: | | also kann man doch auch sagen dass durch PCR1 der nach rechts orientierte Teilstrang bearbeitet wird und durch PCR2 der nach links orientierte teilstrang oder? |
Hast du hier links und rechts verwechselt?
Oder meinst du nicht links und recht von der zu deletierenden (also roten) Sequenz?
| benno67 hat Folgendes geschrieben: | | achso und der überhang von P3 soll bestimmt grün sein oder? |
Soll er.
_________________
RNA?- just another nucleic acid? |
|
 |
benno67
Gast
|
 Verfasst am: 08. Nov 2011 13:55 Titel: Verfasst am: 08. Nov 2011 13:55 Titel: |
 |
|
|
der nach rechts orientierte strang ist bei mir vor PCR1 der gesamte obere teilstrang mit der blauen pfeilspitze und der links orientierte strang ist demnach der andere...stimmt das dann trotzdem?
|
|
 |
jörg

Anmeldungsdatum: 12.12.2010
Beiträge: 2107
Wohnort: Bückeburg
|
 Verfasst am: 08. Nov 2011 14:09 Titel: Verfasst am: 08. Nov 2011 14:09 Titel: |
 |
|
Ich kann nicht so ganz nachvollziehen, was du meinst, befürchte aber, dass du dich da verhaspelt hast:
Mit PCR1 wird der grüne Bereich beider Stränge mit einem blauen Überhang amplifiziert (also links von der zu deletierenden, also roten Sequenz ist) und mit PCR2 der blaue Bereich beider stränge mit einem grünen Überhang (also rechts von der roten Sequenz).
_________________
RNA?- just another nucleic acid? |
|
 |
benno67
Gast
|
 Verfasst am: 08. Nov 2011 14:20 Titel: Verfasst am: 08. Nov 2011 14:20 Titel: |
 |
|
|
am oberen teilstrang kann ich das nachvollziehen wie der grüne bereich mit blauem überhang synthetisiert wird. aber am unteren nicht. P2 kann doch da nicht an der grünen sequenz(und vor der roten) binden...weil dann wären ja primer und ausgangsstrang gleich orientiert und das geht ja nicht?
|
|
 |
jörg

Anmeldungsdatum: 12.12.2010
Beiträge: 2107
Wohnort: Bückeburg
|
 Verfasst am: 08. Nov 2011 15:16 Titel: Verfasst am: 08. Nov 2011 15:16 Titel: |
 |
|
Nee, aber mit P1 wird da ja ein strang synthetisiert, an dem wiederum P2 binden kann.
Ehrlich gesagt verstehe ich auch deine Gedankengänge oftmals nicht.
Malst du dir das wirklich auf und beschriftest die Orientierung der Stränge (also 5'-3' bzw. 3'-5')???
Also nocheinmal:
Im ersten Zyklus bindet an den unteren Strang P1 und die Pol synthetisiert über die rote Sequenz hinaus, meinetwegen bis in den blauen Bereich hinein. Das Ergebnis nennen wir einmal Strang X.
An den oberen Strang bindet P2 und die Pol synthetisiert über die Stelle hinaus, an der P1 bindet. Das Ergebnis sei Strang Y.
Im zweiten Zyklus bindet nun an Strang X P2 und die Pol synthetisiert und an Strang Y bindet P1.
Jeder der beiden Stränge kann nun nur noch bis zu der Stelle synthetisiert werden, an der im vorherigen Zyklus der jeweilige Primer gebunden hat, weil da der Strang zuende ist.
_________________
RNA?- just another nucleic acid? |
|
 |
benno67
Gast
|
 Verfasst am: 08. Nov 2011 15:40 Titel: Verfasst am: 08. Nov 2011 15:40 Titel: |
 |
|
ja ich weiß meine gedankengänge sind nicht immer die einfachsten...aber ich zeichne mir das alles immer auf aber anscheinend nicht ganz so richtig... 
naja...also ich versuch das nochmal so zu beschreiben wie ich es verstanden habe:
unterer strang:
P1 bindet am 3'-ende und polymerase synthetisiert den kompletten komplementären strang also auch über die zu deletierende sequenz hinweg. dann ist ein neuer doppelstrang gebildet. an den komplementären strang bindet P2 vor der roten sequenz und polymerase synthetisiert bis zu der stelle an der P1 gebundet hat, weil ja da der strang zu ende ist. so und dann erhält man ja eben den komplementären strang an dem ein kürzerer strang bindet (von P2 ausgehend bis ehemalige bindungsstelle von P1). kann ich mir das so vorstellen? und dann kann man ja einen weiteren zyklus durchführen wodurch dann nur die kurze sequenz mit überhang entsteht. das gleiche macht man bei PCR2 aber mit P3 und P4. und dann werden die abschnitte wieder zusammengefügt da die enden durch die überhänge komplementär sind. hab ich das jetzt richtig verstanden?
|
|
 |
jörg

Anmeldungsdatum: 12.12.2010
Beiträge: 2107
Wohnort: Bückeburg
|
 Verfasst am: 08. Nov 2011 15:51 Titel: Verfasst am: 08. Nov 2011 15:51 Titel: |
 |
|
| benno67 hat Folgendes geschrieben: |
P1 bindet am 3'-ende und polymerase synthetisiert den kompletten komplementären strang also auch über die zu deletierende sequenz hinweg. |

| benno67 hat Folgendes geschrieben: | | an den komplementären strang bindet P2 vor der roten sequenz und polymerase synthetisiert bis zu der stelle an der P1 gebundet hat, weil ja da der strang zu ende ist. |
Aber noch nicht im ersten Zyklus. Da wird auch über diese Stelle hinwegsynthetisiert. Das ist nur in der Abbildung nicht mit aufgezeichnet.
Wenn der "Mutter"-Strang da zuende sein sollte, hast du recht, das wäre aber unüblich.
| benno67 hat Folgendes geschrieben: | | so und dann erhält man ja eben den komplementären strang an dem ein kürzerer strang bindet (von P2 ausgehend bis ehemalige bindungsstelle von P1). |
Du meinst als Ergebnis des ersten Zyklus? Dann wäre es soweit korrekt.
Wie gesagt, geht dieser Strang normalerweise aber auch über die Bindungsstelle des Primers hinaus, ist nur hier nicht eingezeichnet.
| benno67 hat Folgendes geschrieben: | | und dann kann man ja einen weiteren zyklus durchführen wodurch dann nur die kurze sequenz mit überhang entsteht. |
Genau und in so 25-30 weiteren Zyklen amplifiziert man die dann exponentiell.
_________________
RNA?- just another nucleic acid? |
|
 |
benno67
Gast
|
 Verfasst am: 08. Nov 2011 16:00 Titel: Verfasst am: 08. Nov 2011 16:00 Titel: |
 |
|
benno67 hat Folgendes geschrieben:
so und dann erhält man ja eben den komplementären strang an dem ein kürzerer strang bindet (von P2 ausgehend bis ehemalige bindungsstelle von P1).
eigentlich meine ich damitim ergebnis des zweiten zyklus. man hat ja dann den im ersten zyklus ausgehend von P1 synthetisierten komplementären strang an den dann komplementär der kürzere strang (von P2 ausgehend bis ehemalige bindungsstelle von P1) gebunden ist. Richtig?
|
|
 |
jörg

Anmeldungsdatum: 12.12.2010
Beiträge: 2107
Wohnort: Bückeburg
|
 Verfasst am: 08. Nov 2011 16:06 Titel: Verfasst am: 08. Nov 2011 16:06 Titel: |
 |
|
Du hast also nur einen Strang betrachtet?
Dann ist das soweit richtig.
An dem anderen Strang geschieht natürlich quasi das Gleiche, nur umgekehrt.
Na endlich.... 
_________________
RNA?- just another nucleic acid? |
|
 |
benno67
Gast
|
 Verfasst am: 08. Nov 2011 16:13 Titel: Verfasst am: 08. Nov 2011 16:13 Titel: |
 |
|
supi! das hat aber echt gedauert! 
und die letzte abbildung versteh ich soweit. nur der letzte schritt: hier ist es ja so dass sch die PCR-Produkte aus PCR2 und PCR1 aneinanderlagern weil die roten sequenzen ja komplementär sind. aber wie werden die stränge dann gleich lang? also wie wird ab der roten sequenz weitersynthetisiert?
|
|
 |
jörg

Anmeldungsdatum: 12.12.2010
Beiträge: 2107
Wohnort: Bückeburg
|
 Verfasst am: 08. Nov 2011 16:16 Titel: Verfasst am: 08. Nov 2011 16:16 Titel: |
 |
|
Na ja, wie bei bisher allen Prinzipien besprochen:
Die komplementären Sequenzen der Stränge lagern sich aneinander und die Polymerase vervollständigt sie.
Mit den flankierenden Primern wird die Zielsequenz inkl. der Insertion dann gezielt amplifiziert.
Schau dir dazu noch einmal die ersten Beiträge an....
_________________
RNA?- just another nucleic acid? |
|
 |
benno67
Gast
|
 Verfasst am: 08. Nov 2011 16:21 Titel: Verfasst am: 08. Nov 2011 16:21 Titel: |
 |
|
ok dann danke für deine hilfe!!! 
|
|
 |
jörg

Anmeldungsdatum: 12.12.2010
Beiträge: 2107
Wohnort: Bückeburg
|
 Verfasst am: 08. Nov 2011 16:25 Titel: Verfasst am: 08. Nov 2011 16:25 Titel: |
 |
|

_________________
RNA?- just another nucleic acid? |
|
 |
benno67
Gast
|
 Verfasst am: 08. Nov 2011 17:48 Titel: Verfasst am: 08. Nov 2011 17:48 Titel: |
 |
|
also einmal muss ich dich noch nerven...und zwar beim enfernen eines dna-abschnittes:
laufen PCR1 und PCR2 eigentlich nacheinander oder gleichzeitig ab? eigentlich könnten die doch auch gleichzeitig ablaufen. dabei bindet dann doch P2 an den oberen strang und pol synthetisiert bis zum ende (durch P1 festgelegt). gleichzeitig bindet P3 am unteren Strang.hier ist aber noch kein ende definiert so dass pol über zielsequenz hinweg synthetisiert. oder ist das quatsch?
|
|
 |
jörg

Anmeldungsdatum: 12.12.2010
Beiträge: 2107
Wohnort: Bückeburg
|
 Verfasst am: 08. Nov 2011 18:15 Titel: Verfasst am: 08. Nov 2011 18:15 Titel: |
 |
|
Na ja, das Problem ist halt, wenn P2 und P3 aneinander binden, dann würden da ja Produkte rauskommen, die nur die Primersequenzen fusioniert hätten. Deswegen macht man das nicht.
Zueinander komplementäre Primer in einer PCR zu verwenden ist immer unschön.
_________________
RNA?- just another nucleic acid? |
|
 |
PaGe
Moderator

Anmeldungsdatum: 19.03.2007
Beiträge: 3549
Wohnort: Hannover
|
 Verfasst am: 08. Nov 2011 21:03 Titel: Verfasst am: 08. Nov 2011 21:03 Titel: |
 |
|
 Ihr seid ja fleißig. Ihr seid ja fleißig.
_________________
Die deutsche Rechtschreibung ist Freeware, du darfst sie kostenlos nutzen. Aber sie ist nicht Open Source, d. h., du darfst sie nicht verändern oder in veränderter Form veröffentlichen. |
|
 |
jörg

Anmeldungsdatum: 12.12.2010
Beiträge: 2107
Wohnort: Bückeburg
|
 Verfasst am: 09. Nov 2011 23:24 Titel: Verfasst am: 09. Nov 2011 23:24 Titel: |
 |
|
| PaGe hat Folgendes geschrieben: |  Ihr seid ja fleißig. Ihr seid ja fleißig. |
Na ja, ich hatte gerade etwas Zeit und sass sowieso die ganze Zeit am Rechner... 
_________________
RNA?- just another nucleic acid? |
|
 |
|