| Autor |
Nachricht |
kabotsch
Gast
|
 Verfasst am: 02. Jan 2012 23:34 Titel: RNA Prozessierung bei Eukaryoten Polyadenylierung Verfasst am: 02. Jan 2012 23:34 Titel: RNA Prozessierung bei Eukaryoten Polyadenylierung |
 |
|
Meine Frage:
Ich habe in meinen Uniunterlagen eine Grafik auf der ein sehr grober Überblick über die Polyadenylierung von RNA Transkripten zu sehen ist.
Dabei ist grob folgendes aufgezeichnet:
[DNA] --Transkription--> [Primärtranskripte*] --Spaltung#--> [RNA&] --Polyadenylierung--> [polyadenylierte Primärtranskripte]
* hier sind versch. lange Primärtranskripte aufgezeichnet
# "durch Endonukleasen"
& ab hier sind alle RNA Stücke gleich lang
Mir ist nicht klar warum die Primärtranskripte zuvor überhaupt unterschiedlich lang sind ?
Meine Ideen:
Ich denke das es daran liegt das die Termination nicht immer gleich abläuft und das Primärtranskript an unterschiedlichen Stellen abgespalten wird bevor die Transkription abgebrochen wird ?! Aber wie wird dies beeinflusst ?
|
|
 |
jörg

Anmeldungsdatum: 12.12.2010
Beiträge: 2107
Wohnort: Bückeburg
|
 Verfasst am: 03. Jan 2012 17:12 Titel: Verfasst am: 03. Jan 2012 17:12 Titel: |
 |
|
Ich kann mit deiner "Abfolge" nur bedingt etwas anfangen.
Zum ersten würde ich den Begriff "Primärtranskript" nur für die prä-mRNA verwenden. Sobald sie polyadenyliert und gespleisst ist, handelt es sich also nicht mehr um das Primärtranskript.
Berücksichtige in deinen Betrachtungen auch, dass alternatives Spleissen und alternative Polyadenylierung aus einem Gen verschiedene Transkripte erzeugen können.
Meinst du mit dem Begriff "Spaltung" die Abspaltung der mRNA am 3'-Ende oder das Spleissen?
| kabotsch hat Folgendes geschrieben: |
Ich denke das es daran liegt das die Termination nicht immer gleich abläuft und das Primärtranskript an unterschiedlichen Stellen abgespalten wird bevor die Transkription abgebrochen wird ?! |
Es ist durchaus richtig, dass nicht immer an derselben Stelle terminiert wird, aber eigentlich ist die mRNA schon vom Transkript abgespalten, sobald die Polymerase die "Cleavage-site" passiert hat, der Vorgang erfolgt also kotranskriptionell.
Die Polymerase elongiert aber noch weit über das 3'-Ende der mRNA hinaus. Bei gleicher Poly(A)-site allerdings wird die RNA auch immer an einer Position in der Nähe (meistens ein CA-Dinukleotid) abgespalten. Und selbst wenn die "Cleavage-site" sich unterscheidet, so besteht der Unterschied nur in wenigen Nukleotiden, das macht sich zum einem kaum bis gar nicht bemerkbar, was die Länge betrifft und schlägt sich zum anderen auch in der vollprozessierten, reifen mRNA nieder.
| kabotsch hat Folgendes geschrieben: | | Aber wie wird dies beeinflusst ? |
Während ein Modell vorschlägt, dass die Dissoziation von Elongationsfaktoren oder die Rekrutierung von Terminationsfaktoren vom Transkriptionskomplex den entscheidenden Mechanismus zur Termination darstellt, postuliert ein anderes Modell, dass das nach Spaltung am 3’-Ende der mRNA nun entstehende freie und ungeschützte 5’-Ende des Transkriptes exonukleatisch abgebaut wird. Sobald die an diesem Abbau beteiligte Exonuklease XRN2 die Pol II erreicht hat, treten die beiden Enzyme in eine Wechselwirkung, die zur Transkriptionstermination führt. Beide Modelle sind jedoch noch nicht ausreichend belegt.
Kannst du das Schema posten, ich kann das aus deinen Angaben nämlich leider nicht rekonstruieren?
_________________
RNA?- just another nucleic acid? |
|
 |
kabotsch
Gast
|
 Verfasst am: 03. Jan 2012 22:09 Titel: graph Verfasst am: 03. Jan 2012 22:09 Titel: graph |
 |
|
Vielen Dank erstmals für die umfangreiche Antwort !
Hier die besagte Grafik:
img6.imagebanana.com/img/khmjbaly/graph.png
|
|
 |
jörg

Anmeldungsdatum: 12.12.2010
Beiträge: 2107
Wohnort: Bückeburg
|
 Verfasst am: 03. Jan 2012 22:32 Titel: Verfasst am: 03. Jan 2012 22:32 Titel: |
 |
|
Auf der Abbildung kommen die unterschiedlichen Längen der Primärtranskripte anscheinend durch die unterschiedlichen Terminationsstellen zustande. Dabei wird durch die Abfolge fälschlicherweise der Eindruck erweckt, dass das alle nacheinander geschieht, dabei passiert das währendeinander.
Damit hättest du mit der folgenden Aussage vollkommen recht:
| kabotsch hat Folgendes geschrieben: |
Ich denke das es daran liegt das die Termination nicht immer gleich abläuft |
Aber gespalten wird die RNA stets an der gleichen Stelle (in der Abbildung als Poly(A)-Stelle bezeichnet, obwohl als Poly(A) Signal eigentlich das AAUAAA-Hexamer bezeichnet wird und das dort die "Cleavage-site" ist; aber die Bezeichnungen sind auch oft in der Fachliteratur inkonsistent).
Damit kannst du folgende Aussage nicht treffen:
| kabotsch hat Folgendes geschrieben: | | und das Primärtranskript an unterschiedlichen Stellen abgespalten wird |
Und wie gesagt: Sobald Polyadenyliert und gespleisst ist, spricht man eigentlich nicht mehr von Primärtranskript. Die Abbildung könnte implizieren, dass noch das Primärtranskript polyadenyliert wird, die anderen Prozessierungsschritte aber erst danach erfolgen. Das ist natürlich Quatsch, die Prozessierungsschritte sind eng aneinander gekoppelt und erfolgen kotranskriptionell. Die Prozessierungsfaktoren stehen dabei in enger räumlicher und funktioneller Beziehung zur C-terminalen Domäne der Polymerase II.
| kabotsch hat Folgendes geschrieben: | | Aber wie wird dies beeinflusst ? |
Dazu habe ich ja bereits Stellung genommen.
_________________
RNA?- just another nucleic acid? |
|
 |
kabotsch
Gast
|
 Verfasst am: 03. Jan 2012 23:02 Titel: Verfasst am: 03. Jan 2012 23:02 Titel: |
 |
|
Vielen Dank für die Antwort! Bin jetzt auch schon ein bisschen tiefer in der Materie und sehe wie grob diese Grafik wirklich ist  . .
| jörg hat Folgendes geschrieben: | | .. mRNA schon vom Transkript abgespalten, sobald die Polymerase die "Cleavage-site" passiert hat, der Vorgang erfolgt also kotranskriptionell... |
Mich würde aber noch brennend interessieren wer und wann an dieser Cleavage-site jetzt das RNA Transkript im Falle eines Eukaryoten schneidet ?
Du schreibst das die RNA Polymerase II die DNA fortlaufend in RNA übersetzt. Somit auch über die "Cleavage-site" der RNA hinweg. Die RNA wird dann durch Endonukleasen - richtig ??? an Cleavage-site gespalten und eine endonuklease baut den nach wie vor synthetisierenden Strang der Polymerase ab. Bis sie die Polymerase erreicht und die Synthese abgebrochen wird. Hoffe ich habe das richtig verstanden ?
Heißt das dann das die "Cleavage-site" eine gewisse konservierte Sequenz ist die bei jedem Gen gleich ist (ist sicher nicht so ?!  ) ? ) ?
|
|
 |
jörg

Anmeldungsdatum: 12.12.2010
Beiträge: 2107
Wohnort: Bückeburg
|
 Verfasst am: 04. Jan 2012 00:46 Titel: Verfasst am: 04. Jan 2012 00:46 Titel: |
 |
|
| kabotsch hat Folgendes geschrieben: |
[...] und eine endonuklease baut den nach wie vor synthetisierenden Strang der Polymerase ab. |
Exonuklease, nicht Endonuklease
| kabotsch hat Folgendes geschrieben: | | Bis sie die Polymerase erreicht und die Synthese abgebrochen wird. Hoffe ich habe das richtig verstanden ? |
Das ist ein Modell. Aber die ganze Wahrheit kann das noch nicht sein, da diese Exonuklease allein nicht in der Lage ist, die Termination einzuleiten. Da müssen noch andere Faktoren eine Rolle spielen. Im Fokus sind da auch verschiedene Faktoren, die mit der 3' Prozessierungsmaschinerie wechselwirken.
| kabotsch hat Folgendes geschrieben: | | Heißt das dann das die "Cleavage-site" eine gewisse konservierte Sequenz ist die bei jedem Gen gleich ist? |
Jap, meistens handelt es sich um ein CA-Dinukleotid ca. 20 Nukleotide stromabwärts des AAUAAA-Hexamers.
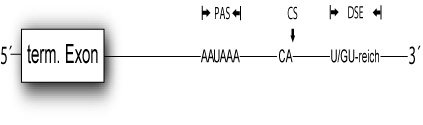
Ich habe dir mal eine Grafik der konservierten Sequenzen angehängt, die bei der 3' Prozessierung eine Bedeutung haben (term. Exon= terminales Exon; PAS= Poly(A) Signal; CS= Cleavage site; DSE= downstream sequence element).
Das PAS wird von einem Faktor namens CPSF (cleavage and polyadenylation specifity factor) erkannt und das DSE von dem Faktor CstF (cleavage stimulation factor). Über diese Wechselwirkung werden dann noch zwei "cleavage Faktoren" rekrutiert. Daraufhin spaltet die Endonuklease CPSF 73 (eine Untereinheit des CPSF) die RNA an der CS. Aber es muss nicht unbedingt ein CA sein, doch in den allermeisten Fällen ist es eines.
Das wäre das vereinfachte Modell, tasächlich sind da noch viel mehr Faktoren beteiligt, aber diese reichen aus, um effektiv zu spalten und sind bei jeder mRNA beteiligt. Die anderen Faktoren unterscheiden sich zwischen den verschiedenen Genen.
| Beschreibung: |
|
| Dateigröße: |
20.16 KB |
| Angeschaut: |
6021 mal |
|
_________________
RNA?- just another nucleic acid? |
|
 |
kabotsch
Gast
|
 Verfasst am: 04. Jan 2012 10:40 Titel: Verfasst am: 04. Jan 2012 10:40 Titel: |
 |
|
|
Danke du hast mir enorm geholfen !
|
|
 |
|